1、方法/步骤1
1、搜索平台基因云馆,登录系统(或注册后登录)。

2、如何进行数据检索、下载、预处理、差异基因分析,详见我们的“解决如何分析GEO数据库中某一疾病的差异基因”的相关文章,这里就略过不表。
通过表达集数据信息 ,可以得到样本的样本数据分组信息(pData),下图:

3、结果是酱紫的:

4、pDatamatrix.csv具体信息:
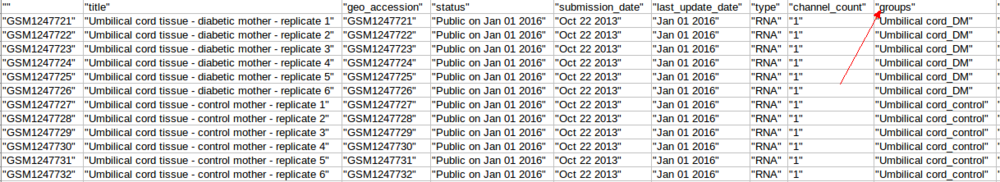
5、其中,groups就是我们的分组。这很重要,分组信息是否正确往往决定了我们能否做出好的分析结果。
通过差异基因分析,可以得到差异基因的表达谱信息,在DEGexp.csv,具体信息:
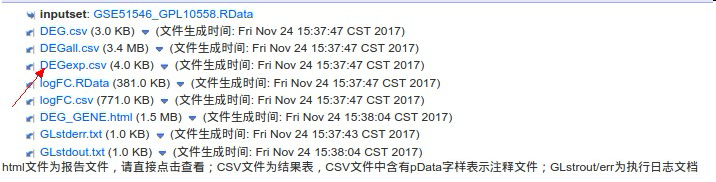
6、点开其中的DEGexp.csv之后,是酱紫的:

7、方法/步骤2
1、热图(heatmap)绘制
在table处输入需要输入绘图的csv文件,这里为DEGexp.csv;在pdata处输入样本数据分组信息pDatamatrix.csv;
colselect处,分组依据参考pDatamatrix.csv文件,填“groups”;
需要对基因数据聚类则在RowCluster处选TRUE,需要对样本数据聚类则在ColCluster处选TRUE,其他的按照默认值,就是这样的:

8、点击运行
运行结束后,结果:
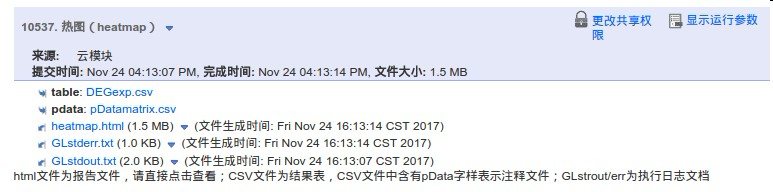
9、点heatmap.html得到下面的样子:

10、发现样本名称有点小,想将字体改大一些,颜色也改动一下,并不对样本数据聚类,该怎么办呢? 往下看“2、热图(heatmap)参数修改”~
2、热图(heatmap)参数修改
点重新运行,Colside填写1.2,则在ColCluster处选FALSE,并在lowcol和highcol处选择想要的颜色:

11、点击运行
运行结束后,结果:
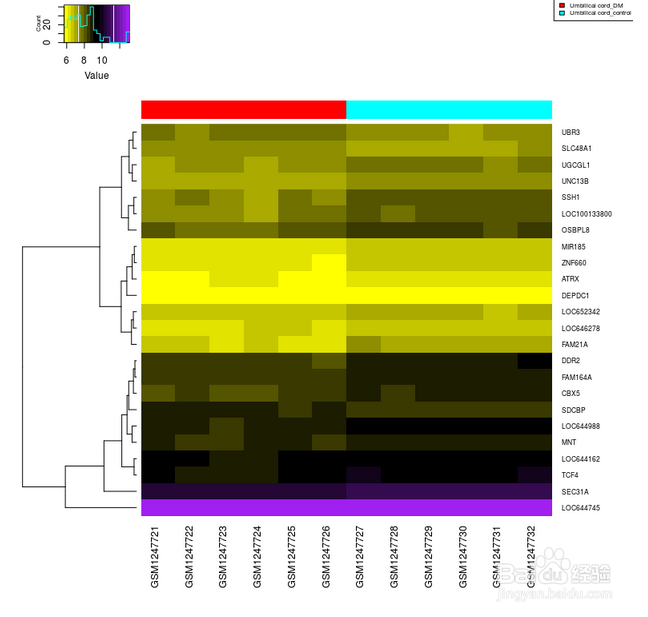
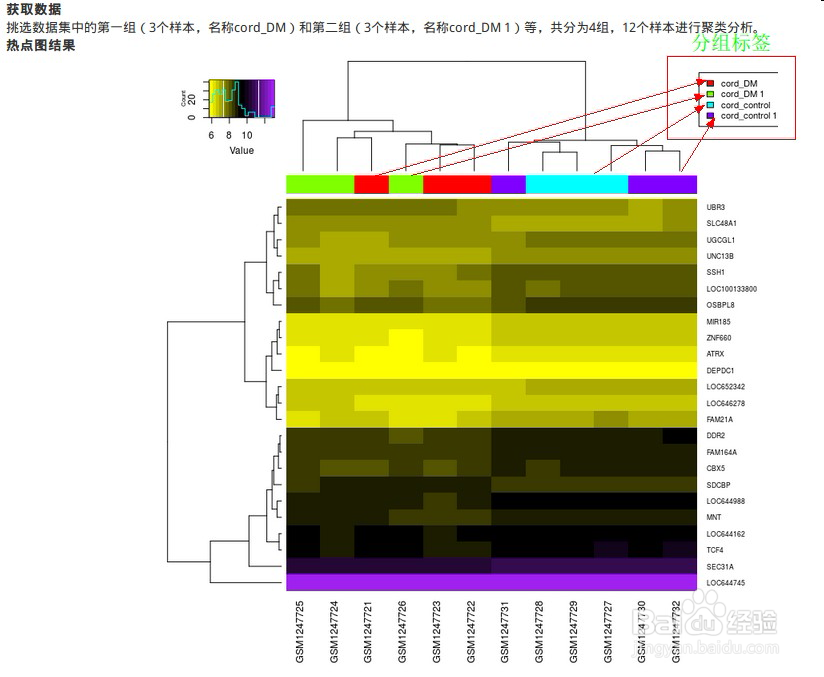
12、疾病数据多个时期热图(heatmap)
很多时候,heatmap 图只能展示出正常组,对照组两组分型的样本分组,遇到多种分型时可怎么办呢?
这个模块的开发者已经为大家考虑到了这种情况,仍然使用这组数据,打开pDatamatrix.csv文件,将分组信息列“groups”数据修改,若疾病数据有4种分型,对应信息如下:
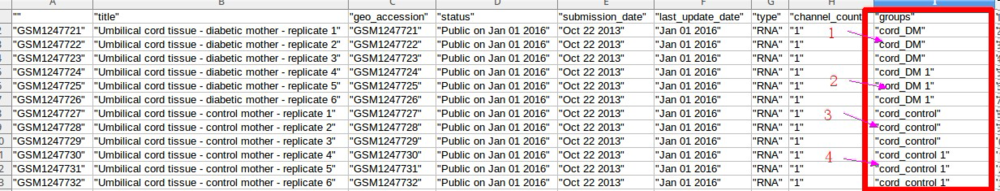
13、(tip:通过修改pDatamatrix.csv文件中的列“groups”,热图中分组标签上的信息是可以随心改动的)并重新上传pDatamatrix.csv文件,按照需求设置好参数,点击运行
运行结束后,结果: